Mit Methoden der künstlichen Intelligenz (KI) gelang es Forscherinnen und Forschern um Professor Dr. Alexander Schönhuth von der Technischen Fakultät der Universität Bielefeld, die Genotypprofile von 3.000 ALS-Patientinnen und -Patienten zu erfassen, aufzuschlüsseln und damit mehr über die Entstehung von ALS zu erfahren. Die Forscher betonen, dass das neue Verfahren es ermögliche, mit 87-prozentiger Genauigkeit zu prognostizieren, ob Personen an ALS erkranken oder nicht.
Bekannt von der Ice Bucket Challenge
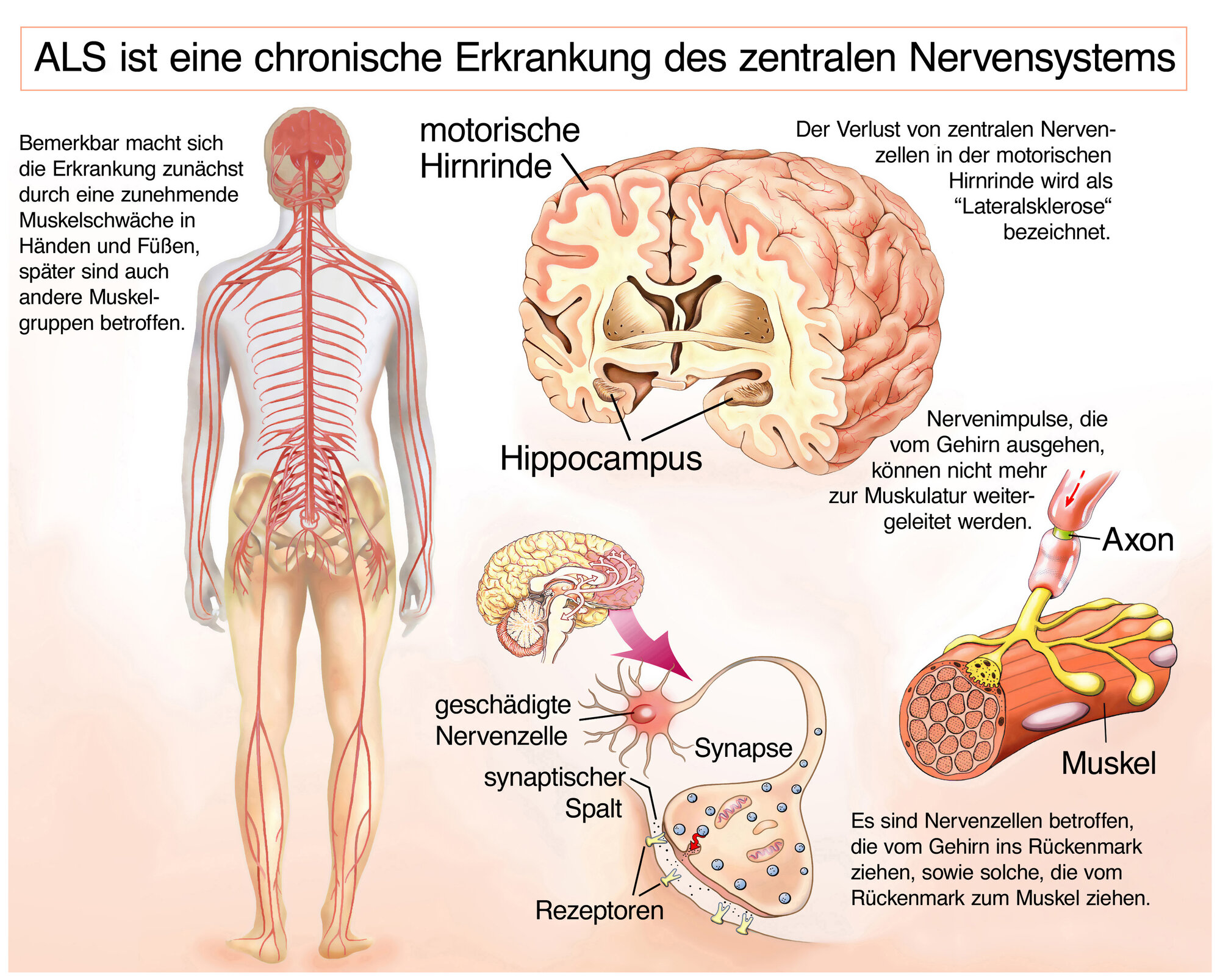
Stephen Hawking ist vielleicht der berühmteste Patient, der an ALS litte. Einer breiten Öffentlichkeit bekannt geworden ist die Krankheit durch die Ice Bucket Challenge, die in den sozialen Medien seinerzeit eine große Aufmerksamkeit erzielte. Insgesamt leiden weltweit circa 150.000 Menschen an der Amyotrophen Lateralsklerose (ALS). Sie wird auch als Motoneuronen-Krankheit bezeichnet. Bei ALS nimmt die Bewegungsfähigkeit der Patientinnen und Patienten im Laufe der Zeit ab und kommt zum Stillstand. Gleichzeitig bleibt die Aktivität des Gehirns komplett funktionsfähig. Der Bioinformatiker Schönhuth entwickelt in seiner Arbeitsgruppe Genome Data Science (Genom-Datenwissenschaft) Methoden und Werkzeuge, um mit Zehntausenden von Genomen zu arbeiten und die Daten zu analysieren. Die Arbeitsgruppe gehört neben der Technischen Fakultät auch zum Centrum für Biotechnologie (CeBiTec) der Universität Bielefeld.
80 Prozent der Erblichkeit bislang unerklärt
„Die Krankheit ALS ist bisher noch in weiten Teilen unverstanden“, sagt Schönhuth. Er hat die Untersuchung zur Diagnose von ALS geleitet. Vor vier Jahren begann er mit der Forschung zu dem Thema, zu der Zeit noch als Arbeitsgruppenleiter am Centrum Wiskunde en Informatica (CWI) in Amsterdam, dem nationalen niederländischen Forschungszentrum für Mathematik und Informatik. „Wir wissen, dass ALS eine vererbbare Krankheit ist, aber 80 Prozent der Erblichkeit sind bislang unerklärt“, sagt der Bioinformatiker. „Bei vielen Erkrankungen, die erblich bedingt sind, gibt es überlappende, sogenannte additive Effekte von genetischen Faktoren – zum Beispiel bei Schizophrenie“, erläutert Schönhuth. „Je mehr dieser Faktoren die Gene aufweisen, um so wahrscheinlicher ist es, dass Personen an Schizophrenie erkranken. Wir können demnach anhand der Gene die genetische Disposition gut erkennen. Bei ALS hingegen ist es viel komplizierter.“ Schönhuth und sein Team nehmen an, dass einzelne Faktoren alleine mit hoher Wahrscheinlichkeit zu ALS führen. Treten diese Faktoren aber gemeinsam auf, ist das Gegenteil der Fall: Es kommt zu keiner Erkrankung. Diese Annahme würde erklären, warum ALS in weiten Teilen unverstanden geblieben ist.
Einsatz von „Capsule Networks“
Die zentrale Methode aus der künstlichen Intelligenz (KI), die Schönhuth und sein Team eingesetzt haben, heißt „Capsule Networks“ (Kapselnetze). Damit wurden genetische Daten von 3.000 ALS-Patienten und 7.000 nicht an ALS-erkrankten Personen ausgewertet. „Der große Vorteil dieses Verfahrens ist, dass Überlappungen von Prozessen erfasst werden können.“ Klassische Methoden kommen mit der Menge an Daten nicht zurecht und liefern keine klaren Ergebnisse. „Unser KI-Verfahren zeigt hingegen nachvollziehbar eindeutig, welche Gene und ihre Prozesse für die Entstehung der ALS-Erkrankung besonders wichtig sind“, sagt Schönhuth.
Sind Medikamente möglich?
Die Ergebnisse der Wissenschaftler/-innen zeigen eine 87-prozentige Genauigkeit im Hinblick auf die Prognose, ob Personen an ALS erkranken oder nicht. „Unser Verfahren kann Vorhersagen bezüglich der Erkrankung treffen. Sie ist viel genauer als andere Methoden. Wir haben mehr als 900 Gene gefunden, die eine Rolle bei der Identifizierung der Erkrankung spielen und 644 Gene, die in komplexen Verbindungen interagieren. Diese Zusammenhänge gilt es in anderen Forschungsgebieten weiter zu untersuchen“, führt Schönhuth aus. „Jedes Gen ist in unterschiedlichen biologischen Prozessen eingebunden. Erfahren wir mehr über die Gene, erfahren wir auch mehr über die Prozesse. So tragen unsere Ergebnisse dazu bei, dass von ALS betroffene Menschen ihren Lebensstil anpassen können, um das Risiko für die Erkrankung zu reduzieren. Zudem könnten auch Medikamente entwickelt werden, die bestimmte Prozesse beeinflussen“, erklärt Schönhuth.
Die aktuelle Studie baut auf Erkenntnissen aus zwei großen Verbundprojekten auf:
- dem internationalen Forschungsnetzwerk „Algorithms for Pangenome Computational Analysis“ (Algorithmen für Berechnungsanalysen des Pangenoms, Kurzname: Alpaca). Die Europäische Union fördert das Netzwerk seit 2020, koordiniert wird es von der Universität Bielefeld.
- dem internationalen Forschungsprojekt „Pan-genome Graph Algorithms and Data Integration“ (Graph-Algorithmen und Datenintegration für Pangenomik, Kurzname: Pangaia). Die Europäische Union fördert das Projekt ebenfalls seit 2020, koordiniert wird es von der Universität Mailand (Italien).
Quelle: idw/Uni Bielefeld
Artikel teilen