TIGER: Transkriptionsgeschichte einzelner Zellen erfahren
Bakterielle und virale Infektionen können schwere akute Beschwerden hervorrufen. Sie können aber auch verheerende langfristige Folgen haben, zum Beispiel eine Krebserkrankung auslösen. Forscherinnen und Forscher suchen deshalb nach neuen Technologien und Ansätzen, um Krankheitsverläufe besser zu verstehen und die Entwicklung von Zellen und Geweben vorherzusagen. Mit immer präziseren Methoden analysieren sie die Prozesse in einzelnen Zellen. So wollen sie beispielsweise eine veränderte Genaktivität nachweisen, die wiederum auf einen krankhaften Prozess hindeuten kann.
Vergangene RNA-Transkripte aufzeichnen
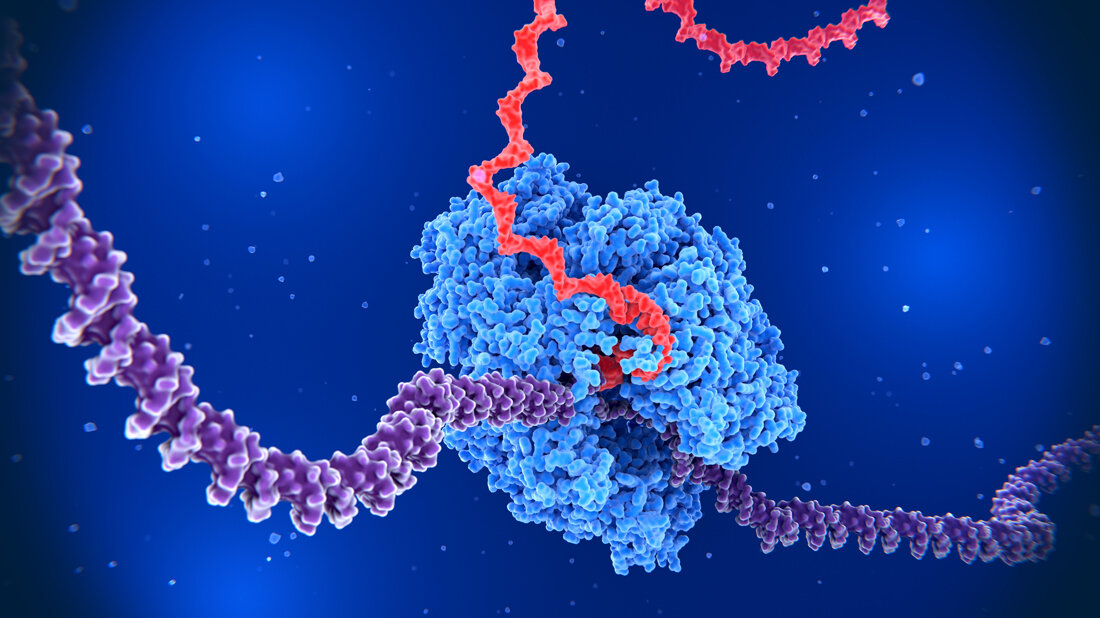
Einen wichtigen Beitrag zum Verständnis leisten dabei Ribonukleinsäuren (RNAs). Sie können Zeugnis darüber geben, welche zellulären Gene „angeschaltet“ sind – denn nur von diesen aktiven Genen werden in der Transkription RNA-Kopien (Transkripte) hergestellt. Eine Herausforderung ist es jedoch, dass die in der Transkription exprimierten RNA-Moleküle nur eine Momentaufnahme darstellen. Vergangene Zellereignisse – beispielsweise eine bakterielle Infektion – mit gegenwärtigen Zuständen zu vernetzen und daraus Folgerungen für die Zukunft abzuleiten, ist durchaus schwierig. Forscherinnen und Forscher des Helmholtz-Instituts für RNA-basierte Infektionsforschung (HIRI) und der Julius-Maximilians-Universität (JMU) in Würzburg haben nun eine neue Technologie namens TIGER („Transcribed RNAs Inferred by Genetically Encoded Records“) entwickelt. Sie soll es ermöglichen, komplexe Prozesse in einzelnen Zellen in vivo zu entschlüsseln, indem sie vergangene RNA-Transkripte aufzeichnet.
Blick in die Vergangenheit
„Die Identität und das Verhalten einer Zelle hängen nicht nur von ihrer aktuellen intrazellulären Beschaffenheit und extrazellulären Umgebung ab, sondern auch von ihren früheren Zuständen. Wir haben nach einem effizienten Verfahren auf Einzelzellebene gesucht, um in die Vergangenheit zu blicken und sie mit der Gegenwart zu verbinden“, erklärt Prof. Chase Beisel. Der Leiter der Abteilung Synthetische RNA-Biologie am Würzburger Helmholtz-Institut für RNA-basierte Infektionsforschung (HIRI) ist leitender Autor der Studie. Das Institut ist ein Standort des Braunschweiger Helmholtz-Zentrums für Infektionsforschung (HZI) in Kooperation mit der Julius-Maximilians-Universität (JMU) Würzburg, an der Beisel auch eine Professur innehat.
Fortschritt für die Diagnostik
In ihrer Publikation stellen die Autorinnen und Autoren einen neuen technologischen Ansatz vor, der die medizinische Diagnostik künftig einen bedeutenden Schritt voranbringen könnte, so die Hoffnungen. TIGER soll es ermöglichen, spezifische Ribonukleinsäuren in einzelnen lebenden Zellen aufzuzeichnen.
„Durch die RNA-Aufzeichnung verbindet TIGER aktuelle Zellzustände mit vergangenen Transkriptionszuständen“, beschreibt Erstautor Chunlei Jiao den technologischen Ansatz. TIGER könne die relative Genexpression, also die zum Ausdruck kommende Genaktivität, quantifizieren, Unterschiede zwischen einzelnen Nukleotiden erkennen, mehrere Transkripte gleichzeitig erfassen und Einzelzellphänomene auslesen.
Übertragung von Antibiotikaresistenzen aufgezeichnet
Für Jiao liegen die Vorzüge des Verfahrens auf der Hand: „An vergangene Zellzustände hat sich die bisherige Forschung mithilfe von riesigen Datenmengen und mathematischen Modellierungen lediglich annähern können, indem sie asynchrone Zellen im Zeitverlauf untersucht hat.“ Mit TIGER konnten die an der Studie beteiligten Wissenschaftler/-innen die Übertragung von Antibiotikaresistenzen zwischen Escherichia-coli-Zellen ebenso aufzeichnen wie die Invasion von Salmonellen in Wirtszellen.
Die Technologie könnte es künftig ermöglichen, im lebenden Organismus in die Transkriptionsgeschichte einzelner Zellen zu blicken und mit dem aktuellen Status quo zu verbinden, um komplexe zelluläre Reaktionen zu entschlüsseln – in vivo und nicht-invasiv. So könne man sich beispielsweise vorstellen, TIGER wie ein Probiotikum einzunehmen, damit der zelluläre Zustand des Verdauungstraktes aufgezeichnet und nach dem Ausscheiden analysiert werde, folgern die Autoren.
Hintergrund:
TIGER setzt reprogrammierte tracrRNAs (Rptrs) ein, um ausgewählte zelluläre Transkripte als gespeicherte DNA-Editierungen in einzelnen lebenden Bakterienzellen aufzuzeichnen. Rptrs sind so konzipiert, dass sie eine Basenpaarung mit erkannten Transkripten eingehen und diese in Leit-RNAs umwandeln. Die Leit-RNAs weisen dann einen Cas9-Baseneditor an, ein eingeführtes DNA-Ziel anzusteuern. Das Ausmaß der Basenbearbeitung kann anschließend durch Sequenzierung abgelesen werden. Die Technologie macht sich Erkenntnisse zunutze, die in einer vorangegangenen Untersuchung zur Entwicklung von LEOPARD, einer Plattform für In vitro-Diagnostik, geführt hat (Jiao C, Sharma S, Dugar G et al., Non-canonical crRNAs derived from host transcripts enable multiplexable RNA detection by Cas9, Science 2021).
Quelle: idw/HZI
Artikel teilen