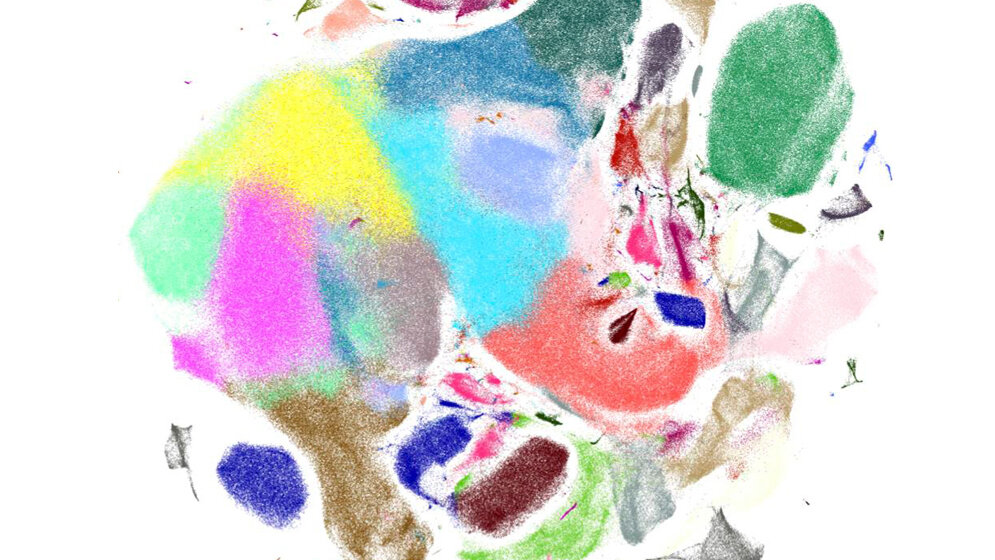„Es geht um die Analyse von Genexpressionsdaten zahlreicher einzelner Zellen“, erklärt Erstautor Alex Wolf vom Institute of Computational Biology (ICB) des Helmholtz Zentrums München. Er hat Scanpy entwickelt, gemeinsam mit seinem Kollegen Philipp Angerer in der Machine Learning Gruppe von Institutsdirektor Prof. Dr. Dr. Fabian Theis, der neben seiner Position am Helmholtz Zentrum auch Professor für Mathematische Modelle biologischer Systeme an der TU München ist. „Die neuen technischen Möglichkeiten generieren um Größenordnungen mehr Daten mit dementsprechend höherer Information“, schildert Theis. „Allerdings war die historisch gewachsene Software-Infrastruktur zur Genexpressionsanalyse nicht auf die neuen Herausforderungen ausgelegt.“ Entsprechend groß sei hier der Bedarf nach neuen Analysemethoden.
Im Rennen für den Human Cell Atlas
Auch ein großes internationales Forschungsvorhaben könnte Theis zufolge von der Software profitieren. Unter dem Namen ‚Human Cell Atlas‘ tragen zahlreiche internationale Wissenschaftler eine Referenzdatenbank zusammen, in der die Genaktivität aller menschlichen Zelltypen erfasst ist. „Für dieses Projekt oder auch bei der immer häufiger werdenden Zusammenlegung von bestehenden Datensätzen ist es wichtig, eine skalierbare Software zu haben“, so Theis. Entsprechend sei Scanpy aktuell in der Auswahl für die Analysesoftware des Human Cell Atlas.
Software wird weltweit eingesetzt
„Mit Scanpy publizieren wir die erste Software, die eine umfängliche Analyse großer Genexpressionsdatensätze mit einem breiten Spektrum aus Methoden des maschinellen Lernens und Statistik erlaubt“, beschreibt Alex Wolf den Fortschritt. „Bereits jetzt wird die Software in diversen Gruppen weltweit eingesetzt, insbesondere auch am Broad Institute von Harvard und dem Massachusetts Institute of Technology.“
Scanpy basiert auf der Sprache Python
Technologisch beschreitet die Anwendung neue Wege: Während entsprechende Biostatistik-Software traditionell in der Programmiersprache R geschrieben wurde, basiert Scanpy auf der Sprache Python, die die Machine Learning Community dominiert. Neu ist zudem, dass Graph-basierte Algorithmen das Herz von Scanpy bilden. Anstatt Zellen wie bisher üblich als Punkte im Koordinatensystem des Genexpressionsraums zu betrachten, verwenden die Algorithmen ein graphartiges Koordinatensystem. Das heißt, anstatt eine Zelle mit dem Expressionswert einiger Tausend Gene zu charakterisieren, wird sie einfach durch die Angabe ihrer nächsten Nachbarn charakterisiert – vergleichbar mit Verbindungen in sozialen Netzwerken. Wenn es zum Beispiel um die Identifikation von Zelltypen geht, verwendet Scanpy also die gleichen Algorithmen wie Facebook zur Identifikation von Communities.
Hintergrund
Das Team und Alex Wolf konnte erst kürzlich einen der vorderen Plätze beim Data Science Bowl belegen, einem der weltweit höchstdotierten Wettbewerbe zum Thema Big Data. In ihrem Beitrag hatte das Team einen Algorithmus programmiert, der binnen weniger Millisekunden Lungenkrebs auf Basis von 300 Schichten eines dreidimensionalen Computertomographie (CT)-Scans erkennt – ein Vorgang für den ein Radiologe im schlechtesten Fall mehrere Stunden benötigen würde. Des Weiteren publizierte das Team kürzlich einen Artikel in Nature Communications zur Rekonstruktion von Zellentwicklung aus Einzelzellbildern: Malen nach Zahlen: Algorithmus rekonstruiert Prozesse aus Einzelbildern. (idw, red)
Wolf, A. et al. (2018): Scanpy: large-scale single-cell gene expression data analysis. Genome Biology, DOI: 10.1186/s13059-017-1382-0.
Artikel teilen